विषयसूची:
- चरण 1: विश्लेषण के लिए छवि का चयन करें और कॉन्फ़िगर करें
- चरण 2: दहलीज और GUI
- चरण 3: प्लॉटिंग कंटूर और सेल डिस्ट्रीब्यूशन
- चरण 4: सेल छवि कनवर्ट करें
- चरण 5: कोशिकाओं की गणना करें और सेल संगम की गणना करें
- चरण 6: कोशिकाओं की गोलाई
वीडियो: BME 60B सैंडबॉक्स प्रोजेक्ट: 6 चरण

2024 लेखक: John Day | [email protected]. अंतिम बार संशोधित: 2024-01-30 09:19
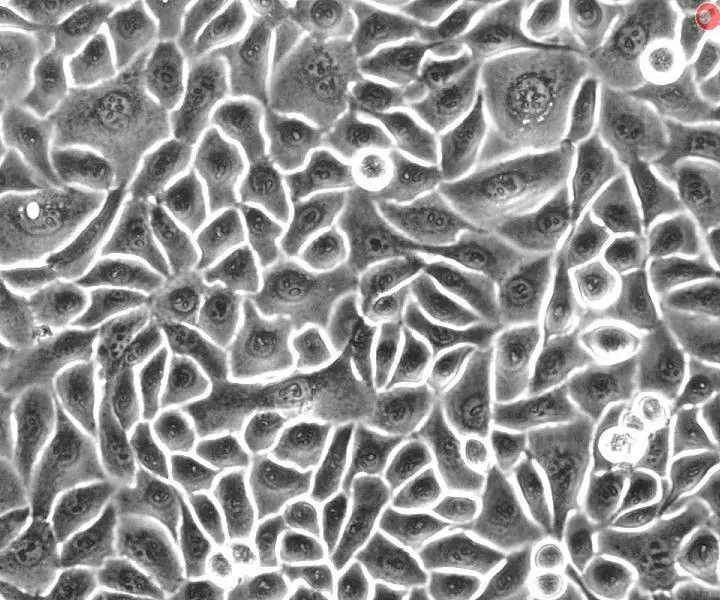
हमारी सैंडबॉक्स परियोजना का उद्देश्य जैविक क्षेत्र में शोधकर्ताओं को कोशिका के नमूनों का विश्लेषण करने और उनकी कोशिकाओं की स्थितियों का पता लगाने में मदद करना है। उपयोगकर्ता द्वारा अपने सेल नमूने की एक छवि इनपुट करने के बाद, हमारा कोड छवि को ग्रेस्केल और बाइनरी में परिवर्तित करके सेल गिनती के लिए तैयार करने के लिए छवि को संसाधित करता है। कोड वास्तविक कोशिकाओं से संबंधित अतिरिक्त जानकारी को हटाने के लिए थ्रेशोल्डिंग का उपयोग करता है ताकि सेल द्रव्यमान को सटीक रूप से खोजा जा सके। छवि में कोशिकाओं की संख्या की गणना करने के बाद, हमारा कोड छवि का पिक्सेल आकार देता है और फिर उपयोगकर्ता को संगम देने के लिए कोशिकाओं की संख्या बनाम कोशिकाओं के क्षेत्र की साजिश रचकर सेल क्षेत्र का वितरण पाता है, जो कि है एक संस्कृति डिश की सतह का प्रतिशत जो ब्याज की कोशिकाओं द्वारा कवर किया जाता है। संगम के आधार पर, शोधकर्ता यह निर्धारित कर सकता है कि कोशिकाओं को पारित किया जाना चाहिए या नहीं; कोशिकाओं के पारित होने का अर्थ है कुछ संस्कृति को एक नए विकास माध्यम में स्थानांतरित करके जीवन या कोशिकाओं या सूक्ष्मजीवों की संख्या का विस्तार करना, और यह तब उपयोगी होता है जब कोशिकाएं बहुत बड़ी हो जाती हैं या भोजन से बाहर हो जाती हैं। कोड चलाने और छवि को संसाधित करने के बाद, उपयोगकर्ता यह तय कर सकता है कि वे परिणामों को स्वीकार करते हैं या अस्वीकार करते हैं, और यदि आवश्यक हो तो डेटा की बेहतर पुनर्प्राप्ति के लिए सीमा को समायोजित करने के लिए आगे बढ़ें।
चरण 1: विश्लेषण के लिए छवि का चयन करें और कॉन्फ़िगर करें
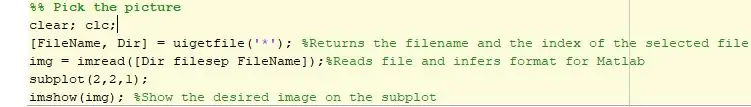
हमारे कोड में पहला कदम उपयुक्त छवि का चयन करना और इसे मैटलैब में विश्लेषण के लिए कॉन्फ़िगर करना है। हम uigetfile फ़ंक्शन का उपयोग करके छवि का चयन करने में सक्षम हैं जो हमें किसी भी फ़ाइल को पकड़ने और प्रोग्राम में डालने की अनुमति देता है। फिर, इमरीड का उपयोग करके हम छवि को पढ़ते हैं और इसे मैटलैब में विश्लेषण के लिए कॉन्फ़िगर करते हैं। तब चयनित छवि को सबप्लॉट पर प्रदर्शित किया जाता है।
चरण 2: दहलीज और GUI
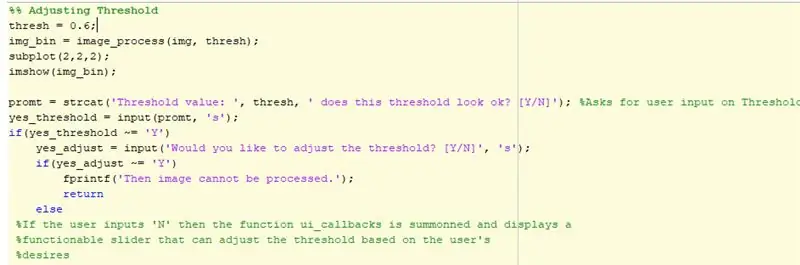
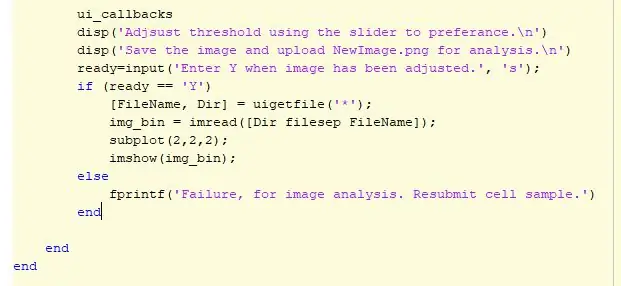
कोड की शुरुआत में "uigetfile" का उपयोग करके एक छवि को चुना जाता है और फिर उस छवि को एक चर के साथ परिभाषित किया जाता है। विभिन्न विश्लेषण चलाते समय कोड की पहचान करने के लिए चर का उपयोग किया जाएगा। चित्र में 2x2 सबप्लॉट बनाया गया है। स्थिति 1 में, मूल छवि प्रदर्शित की जाएगी। कोड का अगला भाग वह जगह है जहां थ्रेसहोल्ड का समायोजन होता है। प्रारंभ में 0.6 की सीमा के डिफ़ॉल्ट का उपयोग किया जाता है और इसे सबप्लॉट की स्थिति 2 में प्रदर्शित किया जाता है। एक if-statement का उपयोग यह निर्धारित करने के लिए किया जाता है कि उपयोगकर्ता थ्रेशोल्ड रखना चाहता है या इसे समायोजित करना चाहता है। उपयोगकर्ता एक जीयूआई का उपयोग करके थ्रेसहोल्ड को समायोजित कर सकता है जिसमें विभिन्न थ्रेसहोल्ड स्थितियों, स्लाइडर और एक सेव बटन पर छवि शामिल है। थ्रेशोल्ड सेट होने के बाद, उपयोगकर्ता छवि को सहेजने के लिए सेव बटन पर क्लिक करेगा और इसे उपयोगकर्ताओं MATLAB फ़ाइलों में-p.webp
चरण 3: प्लॉटिंग कंटूर और सेल डिस्ट्रीब्यूशन
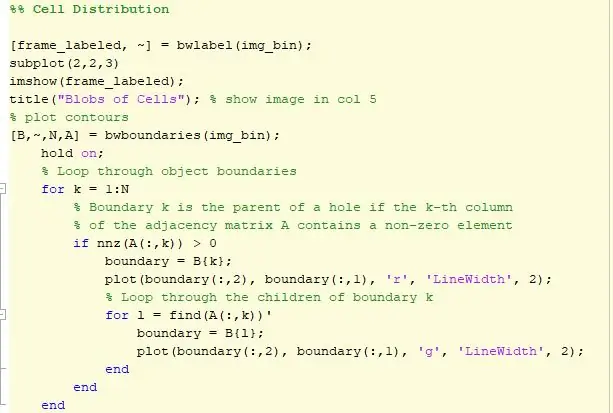
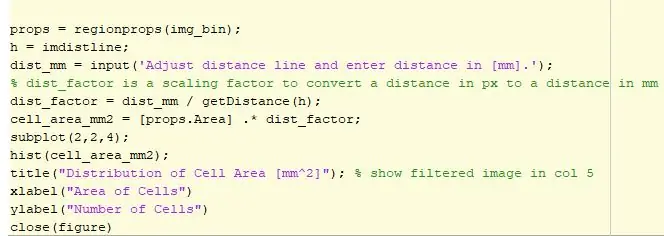
कोड का अगला भाग आकृति को प्लॉट करता है। एक लूप होता है जिसमें कोशिकाएँ एक लाल परिधि से घिरी होती हैं, और वे कोशिकाएँ जो किसी अन्य कोशिका के शीर्ष पर होती हैं, उन्हें हरे रंग में रेखांकित किया जाता है। फिर आउटलाइन इमेज को इंटरेक्टिव डिस्टेंस लाइन के साथ स्थिति 3 में प्रदर्शित किया जाता है। यह रेखा पिक्सेल से मिलीमीटर कनवर्टर के लिए उपयोगकर्ता द्वारा समायोजित लाइन में पिक्सेल की संख्या निर्धारित करेगी। फिर दूरी कारक को क्षेत्रप्रॉप द्वारा निर्धारित क्षेत्र से गुणा किया जाता है और क्षेत्र अब मिलीमीटर वर्ग में व्यक्त किया जाता है। डेटा को उसके क्षेत्र द्वारा कोशिकाओं के वितरण को देखने के लिए हिस्टोग्राम का उपयोग करके प्लॉट किया जाता है। यह हिस्टोग्राम तब स्थिति 4 में प्रदर्शित होगा।
चरण 4: सेल छवि कनवर्ट करें

इस चरण में हमने ग्रेस्केल छवि ली और इसे बिनाराइज़ किया, फ़िल्टर किया और इसे उल्टा कर दिया। छवि पर इन कार्यों को करने से शोर वाले पिक्सेल हटा दिए जाते हैं जो कोशिकाओं के लिए गलत हो सकते हैं और छवि को सेल किनारों के आसपास अधिक चिकनी और नरम बना सकते हैं। यह छवि में कोशिकाओं को अलग-अलग "ब्लॉब्स" के रूप में निकालने के लिए किया गया था जो पृष्ठभूमि से तीव्रता में भिन्न थे। "ब्लब्स" उच्च तीव्रता वाली सफेद छवियां थीं और पृष्ठभूमि काली थी। यदि थोड़ा और समय दिया जाता तो हम अपनी छवियों के लिए अधिक सटीक और अधिक उपयुक्त होने के लिए imbinarize फ़ंक्शन के बजाय एक अलग ब्लॉब छवि रूपांतरण का उपयोग करते, लेकिन हमें फ़ंक्शन को शोध और कार्यान्वित करने के लिए और अधिक समय चाहिए।
चरण 5: कोशिकाओं की गणना करें और सेल संगम की गणना करें

कोड के इस चरण में, हमने छवि में मौजूद कोशिकाओं की संख्या की गणना करने का लक्ष्य रखा है। हमने मुख्य रूप से बूँद के क्षेत्रों की गणना करने के लिए फ़ंक्शन रीजनप्रॉप का उपयोग किया और यदि क्षेत्र की गणना हमारी वांछित सीमा में की जाती है तो इसे एक सबप्लॉट पर प्लॉट किया जाएगा। छोटे शोर वाले पिक्सल या बड़ी तीव्रता को हटाने के लिए सीमाएं निर्धारित की गई थीं जो सेल नहीं थीं। सेल काउंटर तब उन सेंट्रोइड्स की गणना करेगा जो तब प्लॉट किए गए थे और उन्हें लूप के लिए काउंटर में जोड़ देंगे। एक बार कोशिकाओं के क्षेत्र निर्धारित हो जाने के बाद हम संगम की गणना करने में सक्षम थे। कोड में इस चरण का मुख्य महत्व कोशिकाओं के संगम का पता लगाना था और यह कोड के हमारे अंतिम लक्ष्य के लिए महत्वपूर्ण था। हमने प्रत्येक बूँद में पिक्सेल (योग (सभी क्षेत्र)) की गणना करके और फिर इसे छवि के कुल पिक्सेल मान (संख्या (img)) से विभाजित करके गणना की। यह अनुपात हमें संगम देता है और यदि यह ८०% से अधिक होने का निर्धारण किया जाता है तो यह शोधकर्ताओं द्वारा कोशिकाओं को पारित करने का समय है। हमारा लक्ष्य यथासंभव सटीक और सटीक होना था लेकिन सीमित समय के साथ इतनी अशुद्धि हो गई। यदि समय दिया जाता तो हम बूँदों की गिनती को अधिक सटीक बनाने के तरीकों पर ध्यान देते, जैसे कि अधिक फ़िल्टरिंग तकनीक और/या एक हफ़ ट्रांसफ़ॉर्म, क्योंकि उस फ़िल्टरिंग तकनीक को आज़माने के लिए अभी तक पर्याप्त शोध नहीं किया गया था।
चरण 6: कोशिकाओं की गोलाई
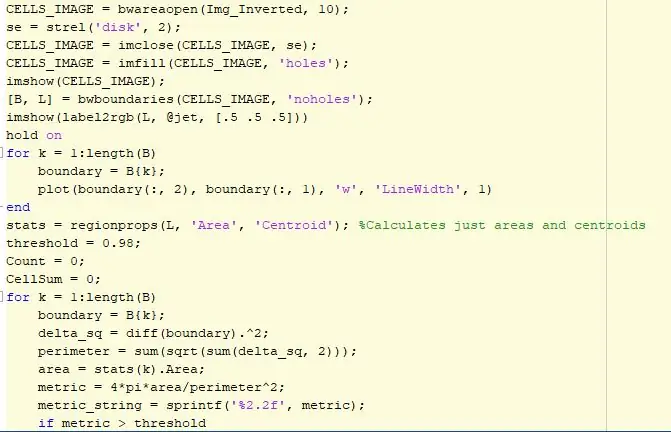

किसी छवि में बूँद की गोलाई को मापने में सक्षम होने से पहले, हमें RGB से ग्रेस्केल में कनवर्ट करने, बिनाराइज़ करने, इनवर्ट करने और छवि को फ़िल्टर करने की आवश्यकता है। एक फ़िल्टरिंग तकनीक bwareaopen फ़ंक्शन का उपयोग कर रही है, जो रुचि की छवि को फ़िल्टर करती है और यह किसी भी ऐसे स्पेक्स या पिक्सेल को हटा देती है जो बहुत छोटे होते हैं जो सेल के आकार का प्रतिनिधित्व नहीं करते हैं। एक संरचना तत्व एक डिस्क के आकार और 2 के पड़ोस के साथ बनाया जाता है और इसका उपयोग पृष्ठभूमि में या कोशिकाओं के भीतर किसी भी अंतराल को भरने के लिए किया जाता है। फिर हम फ़ंक्शन bwboundaries का उपयोग करते हैं जो ब्लॉब्स का पता लगाता है और इसे मैट्रिक्स में संग्रहीत करता है। फिर हम अलग-अलग रंगों का उपयोग करके छवि को लेबल करते हैं ताकि यह एक स्पष्ट दृश्य बन जाए। इसके बाद, लूप के लिए जो छवि में पाई गई वस्तुओं और छिद्रों की संख्या के अनुसार चलता है, यह इस सरणी के अनुरूप बूँद के चारों ओर एक सीमा निर्धारित करता है। एक बार जब यह लूप समाप्त हो जाता है, तो एक और लूप शुरू हो जाता है, फिर से छवि में पाए गए वस्तुओं और छिद्रों की संख्या के अनुसार। इस बार हम फंक्शन रीजनप्रॉप का उपयोग करते हैं जो एरे से क्षेत्र जैसे कुछ गुणों को इकट्ठा करता है और वस्तुओं और छिद्रों की संख्या वाली जानकारी संग्रहीत करता है। इस जानकारी का उपयोग करके, हम केंद्रक आकार का उपयोग करके इन वस्तुओं के क्षेत्रफल और परिधि की गणना करते हैं। एक बार जब हम एक गोलाकार वस्तु की मीट्रिक इकाई की गणना करते हैं और इस माप को सेंट्रोइड्स के बगल में पाठ के रूप में प्लॉट करते हैं, तो परिणामों की तुलना करने के लिए एक सीमा निर्धारित की जाती है। अंतिम कोड छवि में पाए जाने वाले विभिन्न कक्षों की गोलाई प्रदर्शित करेगा और मान के करीब कोई भी मान दूसरों की तुलना में अधिक गोल होगा। 1 के मान का अर्थ है कि सेल पूरी तरह गोल है और पास होने के लिए अच्छी स्थिति में है।
सिफारिश की:
जिम्बल स्टेबलाइजर प्रोजेक्ट: 9 चरण (चित्रों के साथ)

जिम्बल स्टेबलाइजर प्रोजेक्ट: जिम्बल कैसे बनाएंअपने एक्शन कैमरे के लिए 2-एक्सिस जिम्बल बनाना सीखेंआज की संस्कृति में हम सभी को वीडियो रिकॉर्ड करना और क्षणों को कैप्चर करना पसंद है, खासकर जब आप मेरे जैसे कंटेंट क्रिएटर हैं, तो आपने निश्चित रूप से इस मुद्दे का सामना किया है। ऐसा झकझोर देने वाला वीडियो
स्वचालित पेट-फूड बाउल प्रोजेक्ट: १३ चरण

स्वचालित पेट-फ़ूड बाउल प्रोजेक्ट: यह निर्देश योग्य भोजन के कटोरे के साथ एक स्वचालित, प्रोग्राम योग्य पालतू फीडर बनाने का तरीका और वर्णन करेगा। मैंने यहां वीडियो संलग्न किया है जिसमें दर्शाया गया है कि उत्पाद कैसे काम करते हैं और यह कैसा दिखता है
D-882 ट्रांजिस्टर का उपयोग कर शीर्ष 3 बहुत बढ़िया इलेक्ट्रॉनिक्स प्रोजेक्ट: 9 चरण

D-882 ट्रांजिस्टर का उपयोग करते हुए शीर्ष 3 विस्मयकारी इलेक्ट्रॉनिक्स प्रोजेक्ट: JLCPCB चीन में सबसे बड़ा पीसीबी प्रोटोटाइप उद्यम है और एक उच्च तकनीक निर्माता है जो पीसीबी निर्माण के 10 से अधिक वर्षों के अनुभव के साथ त्वरित पीसीबी प्रोटोटाइप और छोटे-बैच पीसीबी उत्पादन में विशेषज्ञता रखता है। वे लागत प्रभावी समाधान प्रदान करने में सक्षम हैं
Arduino Uno के साथ ध्वनिक उत्तोलन चरण-दर-चरण (8-चरण): 8 चरण
Arduino Uno के साथ ध्वनिक उत्तोलन चरण-दर-चरण (8-चरण): अल्ट्रासोनिक ध्वनि ट्रांसड्यूसर L298N डीसी महिला एडाप्टर बिजली की आपूर्ति एक पुरुष डीसी पिन के साथ Arduino UNOBreadboardयह कैसे काम करता है: सबसे पहले, आप Arduino Uno पर कोड अपलोड करते हैं (यह डिजिटल से लैस एक माइक्रोकंट्रोलर है और कोड (C++) कन्वर्ट करने के लिए एनालॉग पोर्ट
सैंडबॉक्स प्रोजेक्ट: बीएसी गणना और व्याख्या: 6 चरण

सैंडबॉक्स प्रोजेक्ट: बीएसी कैलकुलेशन एंड इंटरप्रिटेशन: हारिका गोगिनी, हाना श्लॉसर और बेनेडिक्ट यूसेको द्वारा इस प्रोजेक्ट में, हम किसी विषय के पेय, वजन और लिंग के आधार पर ब्लड अल्कोहल कंसंट्रेशन (बीएसी) की गणना करने का प्रयास करेंगे। परिकलित बीएसी को आउटपुट करने के बाद, हम बताएंगे कि
